Le projet international Human Genome Project (HGP) fut lancé en 1990. Son objectif ultime était de parvenir à décoder le génome humain. «Seuls les meilleurs laboratoires, financés par des pouvoirs publics et chapeautés par une structure internationale, peuvent mener à bien pareil ouvrage titanesque», estimait James Watson, lauréat du prix Nobel et responsable du projet à l’époque (Figure 1). Il avait raison: le séquençage du génome humain allait finalement prendre plus de 10 années et aurait pris plus de temps encore si Craig Venter, à l’époque CEO de la firme de biotechnologies Celera Genomics, ne s’était pas lancé dans la bataille et n’avait pas lancé un projet similaire en 1998. C’est en 2000 que fut présentée, lors d’une conférence de presse commune tenue à la Maison blanche (et organisée consécutivement à des pressions politiques non négligeables), une première ébauche de la séquence du génome humain. La séquence intégrale de l’ADN (3 milliards de paires de bases) de l’Homo sapiens sapiens ne devait finalement être publiée que deux ans plus tard. Coût de l’opération: 3 milliards de dollars US.
Depuis lors, les technologies du séquençage et de l’informatique ont connu une évolution vertigineuse. Au début de cette année, la firme de biotechnologies Illumina Inc. a annoncé qu’elle était à même de décoder l’intégralité du génome humain dans les 24 heures, et ce pour un prix ridicule comparativement aux coûts historiques: 1000 USD (les initiés pensent que ce prix devrait descendre jusqu’à 200 USD dans les années à venir). Cela signifie que le séquençage du génome humain devrait être à la portée de «Monsieur Tout-le-Monde» dans un avenir relativement proche.
| Figure 1: James Watson et Francis Crick. Ils recevront en 1962, conjointement avec Maurice Wilkins, le prix Nobel de physiologie ou médecine pour le décryptage de la structure de l’ADN. |
|---|
 |
Certes, nous connaissons l’ordre des lettres. Mais sommes-nous capables de lire le livre?
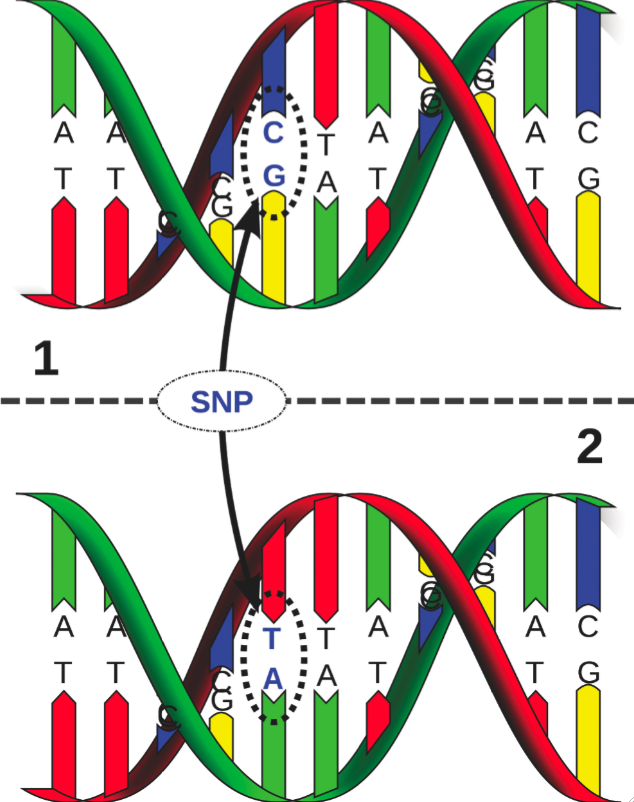
Les (environ) 25.000 gènes renfermés par notre génome sont désormais connus − ce chiffre est nettement inférieur à ce que prévoyaient la plupart des spécialistes il y a dix ans. Depuis lors, l’élucidation de leur(s) fonction(s) fait l’objet d’une activité de recherche scientifique intense. Dans ce contexte, les éléments importants sur un plan clinique sont les polymorphismes d’un seul nucléotide (Single Nucleotide Polymorphisms − ou SNP), à savoir les variations individuelles d’un nucléotide unique au sein de notre ADN (Figure 2). Approximativement 90% de l’ensemble des variations génétiques à l’intérieur du génome humain résident dans des polymorphismes SNP: ce sont eux qui constituent la base génétique de notre identité. Si la contribution d’un SNP est souvent incertaine (la modification de certains nucléotides dans notre ADN ne génère apparemment ni avantage ni inconvénient de type évolutif), les SNP sont, dans certains cas, associés de manière significative à une pathologie. Ces dernières années, la recherche académique et clinique visant à identifier les SNP et leur relation avec certaines affections a connu un essor exponentiel. Les cliniciens et chercheurs estiment que la recherche de ce type contribuera, entre autres, à la mise au point de modes de traitement davantage personnalisés dans un grand nombre de domaines médicaux. Et, interrogé à ce propos, le généticien américain Francis Collins, successeur de James Watson en tant que responsable du projet HGP, d’expliquer que «pour certaines affections, les SNP nous permettent déjà de prédire avec une précision relativement satisfaisante quelles sont les personnes qui présentent un risque accru. Mais soyons clairs: nous n’en sommes qu’au tout début d’une évolution, les avantages cliniques décisifs des analyses de génome doivent encore venir.»
| Figure 2: Les SNP (polymorphismes d’un seul nucléotide) ou combinaisons de SNP sont, dans certains cas, significativement associés à une affection. |
|---|
 |
L’analyse de génome par Internet
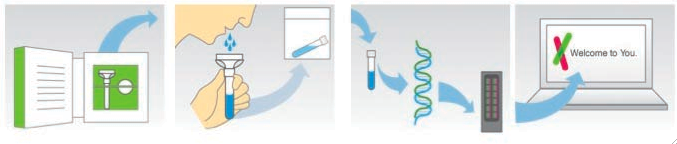
Plusieurs sociétés américaines (23andMe et deCODEme, entre autres) offrent la possibilité de faire procéder à une analyse de génome par Internet pour moins de 100 USD. Les demandeurs peuvent commander un kit par l’intermédiaire du site de la société (www.23andme.com, www.decodeme.com), lequel kit est destiné à recueillir un échantillon de salive et doit ensuite être renvoyé (Figure 3). L’ADN de l’échantillon est alors analysé à l’aide de la technologie DNA-Microarray (développée par Illumina Inc.) sur la présence de 1 million de SNP. Autrement dit, on établit un profil SNP de la personne concernée (certes toujours incomplet, vu que l’on estime qu’environ 10 millions de SNP sont répartis sur l’ensemble du génome humain). L’interprétation des données génétiques peut être demandée une semaine plus tard par l’intermédiaire du site également. Le profil génétique individuel fournit des informations sur:
-
des affections génétiques, telles que la maladie de Huntington ou la mucoviscidose; l’analyse de génome permet de savoir si la personne est porteuse ou non d’un allèle récessif (associé à la pathologie);
-
la réponse individuelle à certaines thérapies: un exemple bien connu à cet égard est celui de la warfarine, un anticoagulant actif exclusivement dans une fenêtre thérapeutique très étroite et qui peut entraîner des hémorragies internes fatales chez certains patients. L’activité de la warfarine est déterminée à 40% (outre d’autres paramètres comme, par exemple, l’âge et la constitution physique de la personne) par deux gènes: la cytochrome P-450-réductase (CYP2C9) et la vitamine K époxyde-réductase (VKORC1). La détermination de la variante SNP permet de prédire la réponse individuelle à l’anticoagulant et de connaître la dose de principe actif de warfarine sûre. Un autre exemple est celui du tamoxifène, un modulateur sélectif du récepteur des oestrogènes mis en oeuvre pour le traitement de certaines formes du cancer du sein. Ce médicament s’avère dépourvu de tout effet thérapeutique chez environ 6% des patientes à cause d’un polymorphisme au niveau du gène cytochrome P450 3A4 (CYP3A4). Une analyse de génome permet de connaître la sous-population à laquelle la personne appartient;
-
profil de risque pour une série d’affections, telles que le diabète de type 2, le cancer du sein, le cancer du colon, les affections cardiovasculaires et vasculaires cérébrales, la dégénérescence de la macula liée à l’âge, ainsi que la maladie d’Alzheimer.
| Figure 3: Analyse de génome par Internet. Le demandeur commande un kit avec lequel un échantillon de salive est prélevé. L’ADN de cet échantillon est analysé sur la présence de 1 million de SNP. Il est ensuite possible de demander à obtenir son profil de risque pour une série d’affections. |
|---|
 |
Il apparaîtra clairement à tout un chacun que les analyses de génome individuelles peuvent contribuer au développement de thérapies plus personnalisées, en ce que les données génétiques d’une personne viennent (ou viendraient) alors prendre leur place dans le profil de santé de la personne. Mais qu’en est-il réellement? Un nombre non négligeable de spécialistes estiment que cette médecine bio-informatisée fait de l’analyse d’ADN le point central de la démarche diagnostique et thérapeutique en ignorant l’ensemble des autres paramètres cliniques pertinents.
Un profil de risque en un clic?
Une question centrale dans ce débat est la suivante: quelle est la crédibilité d’analyses de génome proposées par Internet? Est-il réellement possible de dresser un profil de risque fiable à l’aide d’une combinaison déterminée de SNP pour une affection donnée? Les avis sont partagés. Bon nombre d’affections sont en effet multifactorielles et la composante génétique (lorsqu’elle existe) n’est pas la seule à intervenir: les facteurs environnementaux et le mode de vie jouent également un rôle dans la survenue ou la non-survenue d’une maladie. Un exemple typique est celui du diabète de type 2, une affection qui, outre la prédisposition génétique, est déterminée par des paramètres cliniques fortement associés au mode de vie, comme l’obésité, le profil alimentaire, la consommation d’alcool et le niveau d’activité physique. De fait, la valeur prédictive de ces paramètres (impossible à exciper d’une analyse ADN) en vue de l’évaluation du risque de diabète de type 2 est actuellement plus précise qu’une combinaison donnée de SNP. Il en va de même pour beaucoup d’affections cardiovasculaires et un grand nombre d’autres maladies. Pour énoncer les choses simplement: c’est la relation entre nos «empreintes digitales» génétiques et les facteurs environnementaux qui est déterminante pour notre profil de santé. Un pronostic de risque reposant exclusivement sur une analyse ADN est dès lors, aux yeux de nombreux spécialistes, peu significatif.
Le Pr ém. Uta Francke (Genetics & Pediatrics Dept., Stanford Medical School et Senior Medical Director chez 23andMe) ne conteste nullement que les estimations en matière de risque soient encore imprécises pour un très grand nombre d’affections. «Mais», explique-t-elle, «il est bel et bien possible à l’heure actuelle de dresser un profil de risque acceptable pour toute une série d’affections (23andMe établit, à ce jour, des profils de risque pour 97 affections et deCODEme pour 47).» Uta Francke souligne par ailleurs que les pronostics sont appelés à devenir de plus en plus précis à l’avenir, et ce pour les raisons suivantes: tout d’abord, la base de données de 23andMe renferme actuellement 125 000 analyses de génome individuelles, un nombre qui s’accroît de jour en jour. Ces données sont constamment évaluées au regard de la littérature biomédicale la plus récente et permettent donc de mettre en évidence de nouvelles associations entre génotypage (comme les combinaisons de SNP) et certains profils de risque. Les prédictions deviendront de plus en plus précises au fur et à mesure de l’enrichissement des bases de données. Les personnes testées peuvent d’ailleurs demander régulièrement une mise à jour de leur profil de risque par l’intermédiaire du site. Ensuite, 23anMe réalise des enquêtes par Internet auprès des personnes testées en vue d’obtenir des informations concernant (entre autres) leur mode de vie, leur constitution physique, leurs habitudes alimentaires, la présence d’affections à caractère familial, ainsi que leur état de santé général. La société vise par là à obtenir une image aussi précise que possible de l’ensemble des paramètres cliniques pertinents. Plus de 70% des personnes testées ont rempli ces questionnaires et ont consenti à ce que leurs données soient utilisées (de manière anonyme) à des fins scientifiques. Ce qui a débouché sur des résultats: l’an passé, notamment, a paru une publication de l’équipe scientifique de 23andMe dans laquelle deux nouveaux locus significativement associés à la maladie de Parkinson étaient identifiés (Do C.B. et al., PloS Genetics − juin 2011).
Intérêt des firmes pharmaceutiques
Uta Francke souligne en outre que la firme de biotechnologies 23andMe n’exclut pas de collaborer avec l’industrie pharmaceutique à l’avenir. De fait, le diagnostic génétique joue un rôle non négligeable dans le développement de certains médicaments et codétermine la réussite d’une thérapie (exemples: le cétuximab pour le traitement du cancer colorectal, le trastuzumab pour le cancer du sein et l’imatinib pour la prise en charge de la leucémie myéloïde chronique). Plusieurs firmes pharmaceutiques sont dès lors très intéressées par le fait de disposer de profils ADN individuels qui − élément important − soient représentatifs à l’échelle d’une vaste population randomisée.
Que faire des données?
Une analyse de génome telle que proposée par 23andMe et deCODEme permet à une personne de connaître par Internet son pourcentage de risque concernant l’une ou l’autre maladie. Or, ces données − notons qu’il s’agit, dans certains cas, d’affections graves partiellement mortelles − ne font l’objet d’aucune interprétation par une personne spécialisée ou un médecin traitant. Clairement, cela peut dans certains cas (p.ex. dans le cas d’un risque très élevé de cancer) être source d’angoisse et de stress chez certaines personnes. Ces pronostics peuvent en outre influencer des gens dans toute une série de choix de vie. Une personne qui a été identifiée comme porteuse d’un gène mutant responsable d’une anomalie congénitale sévère (une fois encore: en dehors de tout encadrement médical) va-t-elle envisager différemment les questions du mariage ou de la conception d’enfants? Une personne présentant un risque d’Alzheimer très élevé (maladie pour laquelle aucun traitement adéquat n’a encore pu être mis au point) va-t-elle ressentir ce pronostic comme une condamnation à mort différée? Plusieurs études réalisées aux États-Unis (où ce débat a cours actuellement) s’attachent à apporter des éclaircissements sur ces points. Le risque de voir certaines personnes sur-interpréter leur profil de risque et le confondre avec un pronostic pour l’avenir n’est en effet pas imaginaire. D’autre part, une confrontation avec le profil personnel peut également offrir des avantages. Francis Collins signalait dans une interview récente que son analyse d’ADN avait révélé la présence d’un risque élevé pour le diabète de type 2. «Cela m’a secoué et réveillé», explique-t-il, «j’ai modifié mon régime alimentaire et je me suis mis à faire du sport de manière rigoureuse et disciplinée. J’ai récupéré mon poids optimal et je me sens mieux que jamais auparavant.» On peut en effet envisager des scénarios dans lesquels une personne présentant un risque élevé de cancer colorectal se soumettrait régulièrement à une colonoscopie et se ferait enlever des polypes à titre préventif avant que ceux-ci n’évoluent (potentiellement) vers la malignité.
Des défis pour l’avenir
Il est communément admis dans les milieux médicaux que le diagnostic et le traitement thérapeutique sont appelés à se dérouler de manière plus personnalisée à l’avenir. Les tests génétiques (déjà mis en oeuvre dans certains cas) gagneront en importance dans certains domaines cliniques dans la perspective d’une caractérisation univoque du patient. Peu mettent en doute le fait que les analyses de génome (séquençage complet de l’ADN ou caractérisation de SNP) puissent, elles aussi, contribuer dans certains cas à l’établissement d’un diagnostic ou à la définition de la thérapie optimale. Certains experts pensent cependant que les analyses de génomes sont vouées à devenir des procédures de routine pratiquées pour déterminer le profil de santé et de risque individuel. Quoi qu’il en soit, Uta Francke se montre résolument affirmative: «Dans le futur, dans les pays développés, la plupart des gens connaîtront leur séquence de génome complète. J’en ai la conviction. Peut-être même le séquençage du génome sera-t-il réalisé systématiquement à la naissance d’un enfant.» On peut imaginer en effet qu’à l’avenir, les gens portent une puce renfermant leur séquence ADN individuelle, ce qui permettrait au médecin traitant de prendre la bonne décision thérapeutique en cas d’urgence, pour déterminer la dose de warfarine active sûre, par exemple.
Il va cependant de soi que la pratique systématique d’un séquençage du génome et l’utilisation de telles informations médicales soulèvent des questions sur le plan éthique, voire juridique. Il n’est pas absurde de penser qu’au vu de l’augmentation constante des coûts en matière de soins de santé, l’assurance maladie exige les résultats d’une analyse de génome préalablement à l’établissement d’une police d’assurance adaptée. D’autre part, les médecins pourront-ils encore justifier, d’un point de vue déontologique, la pratique d’interventions médicales très onéreuses et/ou l’instauration de thérapies exclusives chez des patients dont l’analyse ADN révèle un profil de risque négatif et cliniquement pertinent dans le contexte donné? Il semble en tout cas plausible que les résultats de l’analyse de génome soient, à l’avenir, inclus dans le processus de décision. Et que fera-t-on si de futurs parents demandent un séquençage de génome prénatal? À l’heure actuelle, cette procédure ne pose aucun problème insurmontable sur le plan technologique. Quels seront les profils de risque qui justifieront une interruption de grossesse (ou la non-implantation d’un ovule fécondé)? Quelles que soient les opinions des uns et des autres concernant la popularisation des analyses de génome, de plus en plus de voix se font entendre aux USA en faveur de la tenue d’un débat éthique global et de plus de clarté sur le plan juridique. Ces mêmes questions se poseront également en Europe. Ce n’est qu’une question de temps.